Журнал «Здоровье ребенка» Том 16, №2, 2021
Вернуться к номеру
Біогенез мікроРНК. Частина 1. Матурація пре-мікроРНК. Матурація канонічних мікроРНК
Авторы: Абатуров О.Є., Бабич В.Л.
Дніпровський державний медичний університет, м. Дніпро, Україна
Рубрики: Педиатрия/Неонатология
Разделы: Справочник специалиста
Версия для печати
У науковому огляді наведено біогенез мікроРНК. Для написання статті здійснювався пошук інформації з використанням баз даних Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka. У статті подана коротка характеристика послідовності РНК, що кодують мікроРНК. Підкреслено, що мікроРНК залежно від розташування послідовності, що кодує їх в геномі, розподілені на дві великі групи: канонічні і неканонічні miR. Встановлено, що один локус послідовності, що кодує мікроРНК, може генерувати серію некодуючих зрілих транскриптів. Виділяють канонічний та неканонічний (альтернативний) шляхи матурації пре-мікроРНК. Канонічний шлях матурації мікроРНК обумовлений функціонуванням протеїнів DROSHA і DICER. Розкрито внутрішньоядерний процесинг пре-мікроРНК протеїном DROSHA, що призводить до утворення пре-мікроРНК, які транспортуються з ядра клітини в цитоплазму, де під впливом протеїну DICER перетворюються в дуплексні мікроРНК. Дуплексні мікроРНК рекрутуються протеїнами Argonaute (AGO), на яких вони розкручуються, в результаті чого одна з двох ниток РНК стає зрілою мікроРНК. Неканонічні первинні транскрипти мікроРНК можуть піддаватися DROSHA-, DGCR8-незалежному і DICER-незалежному процесингу. Порушення функціонування протеїнів мікропроцесора та ядерного експорту пре-мікроРНК супроводжується розвитком деяких захворювань людини. Таким чином, у біогенезі мікроРНК виділяють канонічний та неканонічний (альтернативний) шляхи матурації пре-мікроРНК. Канонічний шлях матурації первинних транскриптів мікроРНК обумовлений функціонуванням протеїнів DROSHA і DICER. Неканонічний шлях матурації пре-мікроРНК виконується за рахунок DROSHA-, DGCR8-незалежного і DICER-незалежного процесингу. Порушення функціонування різних механізмів канонічного шляху матурації пре-мікроРНК пов’язане з розвитком деяких захворювань людини.
The scientific review presents the biogenesis of miRNAs. To write the article, information was searched using databases Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka. The article presents a brief description of the RNA sequence encoding miRNAs. It is emphasized that microRNAs, depending on the location of the sequence encoding them in the genome, are divided into two major groups: canonical and non-canonical miRNAs. It has been found that a single locus of a sequence encoding a miRNA can generate a series of non-coding mature transcripts. It is noted that there are canonical and non-canonical (alternative) ways of maturation of pri-miRNAs. The canonical path of maturation of miRNAs results from the functioning of DROSHA and DICER proteins. Intranuclear processing of pri-miRNA by the DROSHA protein is revealed, which leads to the formation of pre-miRNAs transported from the cell nucleus to the cytoplasm, where under the influence of the DICER protein they are converted into duplex microRNAs. Duplex miRNAs are recruited by Argonaute (AGO) proteins, on which they are spun, and as a result one of the two strands of RNA becomes mature miRNA. Non-canonical primary miRNA transcripts can be subjected to DROSHA-, DGCR8-independent, and DICER-independent processing. The dysfunction of microprocessor proteins and nuclear export of pre-miRNAs is accompanied by the development of some human diseases. Thus, in the biogenesis of miRNAs, there are canonical and non-canonical (alternative) ways of maturation of pri-miRNAs. The canonical path of maturation of primary microRNA transcripts is due to the functioning of DROSHA and DICER proteins. The non-canonical path of maturation of pre-miRNAs is performed by DROSHA-, DGCR8-independent, and DICER-independent processing. The dysfunction of various mechanisms of the canonical path of maturation of pre-miRNA is associated with the development of some human diseases.
мікроРНК; матурація пре-мікроРНК; протеїни Argonaute (AGO); огляд
microRNA; miRNA; maturation of prі-miRNA; Argonaute (AGO) proteins; review
Вступ
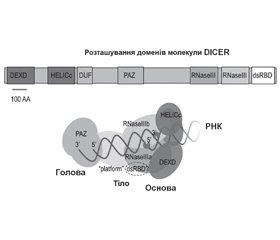
Матурація пре-мікроРНК
Висновки
- Auyeung V.C., Ulitsky I., McGeary S.E., Bartel D.P. Beyond secondary structure: primary-sequence determinants license pri-miRNA hairpins for processing. Cell. 2013 Feb 14. 152(4). 844-58. doi: 10.1016/j.cell.2013.01.031.
- Bartel D.P. Metazoan MicroRNAs. Cell. 2018 Mar 22. 173(1). 20-51. doi: 10.1016/j.cell.2018.03.006.
- Bernstein E., Kim S.Y., Carmell M.A. et al. Dicer is essential for mouse development. Nat. Genet. 2003 Nov. 35(3). 215-7. Doi: 10.1038/ng1253.
- Braun D.A., Sadowski C.E., Kohl S. et al. Mutations in nuclear pore genes NUP93, NUP205 and XPO5 cause steroid-resistant nephrotic syndrome. Nat. Genet. 2016 Apr. 48(4). 457-65. doi: 10.1038/ng.3512.
- Chang T.C., Pertea M., Lee S. et al. Genome-wide annotation of microRNA primary transcript structures reveals novel regulatory mechanisms. Genome Res. 2015 Sep. 25(9). 1401-9. doi: 10.1101/gr.193607.115.
- Chen Y., Wang X. miRDB: an online database for prediction of functional microRNA targets. Nucleic Acids Research. 2020. 48(D1). D127-D131. https://doi.org/10.1093/nar/gkz757.
- Chen Z., Wu J., Yang С. et al. DiGeorge syndrome critical region 8 (DGCR8) protein-mediated microRNA biogenesis is essential for vascular smooth muscle cell development in mice. J. Biol. Chem. 2012 Jun 1. 287(23). 19018-28. doi: 10.1074/jbc.M112.351791.
- Creugny A., Fender A., Pfeffer S. Regulation of primary-microRNA processing. FEBS Lett. 2018 Apr 23. doi: 10.1002/1873-3468.13067.
- Daniels S.M., Melendez-Peña C.E., Scarborough R.J. et al. Characterization of the TRBP domain required for dicer interaction and function in RNA interference. BMC Mol. Biol. 2009 May 7. 10. 38. doi: 10.1186/1471-2199-10-38.
- Daniels S.M., Gatignol A. The multiple functions of TRBP, at the hub of cell responses to viruses, stress, and cancer. Microbiol. Mol. Biol. Rev. 2012 Sep. 76(3). 652-66. doi: 10.1128/MMBR.00012-12.
- Daugaard I., Hansen T.B. Biogenesis and Function of Ago-Associated RNAs. Trends Genet. 2017 Mar. 33(3). 208-219. doi: 10.1016/j.tig.2017.01.003.
- Desvignes T., Batzel P., Berezikov E. et al. miRNA Nomenclature: A View Incorporating Genetic Origins, Biosynthetic Pathways, and Sequence Variants. Trends Genet. 2015 Nov. 31(11). 613-626. doi: 10.1016/j.tig.2015.09.002.
- Fang W., Bartel D.P. The Menu of Features that Define Primary MicroRNAs and Enable De Novo Design of MicroRNA Genes. Mol. Cell. 2015 Oct 1. 60(1). 131-45. doi: 10.1016/j.molcel.2015.08.015.
- Graves P., Zeng Y. Biogenesis of mammalian microRNAs: a global view. Genomics Proteomics Bioinformatics. 2012 Oct. 10(5). 239-45. doi: 10.1016/j.gpb.2012.06.004.
- Guo L., Chen F. A challenge for miRNA: multiple isomiRs in miRNAomics. Gene. 2014 Jul 1. 544(1). 1-7. doi: 10.1016/j.gene.2014.04.039.
- Ha M., Kim V.N. Regulation of microRNA biogenesis. Nat. Rev. Mol. Cell. Biol. 2014 Aug. 15(8). 509-24. doi: 10.1038/nrm3838.
- Hand N.J., Master Z.R., Lay J. Le, Friedman J.R. Hepatic function is preserved in the absence of mature microRNAs. Hepatology. 2009 Feb. 49(2). 618-26. doi: 10.1002/hep.22656.
- Heyam A., Lagos D., Plevin M. Dissecting the roles of TRBP and PACT in double-stranded RNA recognition and processing of noncoding RNAs. Wiley Interdiscip. Rev. RNA. 2015 May-Jun. 6(3). 271-89. doi: 10.1002/wrna.1272.
- Joshua-Tor L. The Argonautes. Cold Spring Harb. Symp. Quant. Biol. 2006. 71. 67-72. Doi: 10.1101/sqb.2006.71.048.
- Kim J.O., Bae J., Kim J. et al. Association of MicroRNA Biogenesis Genes Polymorphisms with Ischemic Stroke Susceptibility and Post-Stroke Mortality. J. Stroke. 2018 Jan. 20(1). 110-121. doi: 10.5853/jos.2017.02586.
- King V.M., Borchert G.M. MicroRNA Expression: Protein Participants in MicroRNA Regulation. Methods Mol. Biol. 2017. 1617. 27-37. doi: 10.1007/978-1-4939-7046-9_2.
- Leisegang M.S., Martin R., Ramírez A.S. et al. Exportin t and Exportin 5: tRNA and miRNA biogenesis- and beyond. Biol. Chem. 2012 Jul. 393(7). 599-604. doi: 10.1515/hsz-2012-0146.
- Li J., Chen Y., Qin X. et al. MiR-138 downregulates miRNA processing in HeLa cells by targeting RMND5A and decreasing Exportin-5 stability. Nucleic Acids Res. 2014 Jan. 42(1). 458-74. doi: 10.1093/nar/gkt839.
- Li S., Patel D.J. Drosha and Dicer: Slicers cut from the same cloth. Cell. Res. 2016 May. 26(5). 511-2. doi: 10.1038/cr.2016.19.
- Liu H., Liang C., Kollipara R.K. et al. HP1BP3, a Chromatin Retention Factor for Co-transcriptional MicroRNA Processing. Mol. Cell. 2016 Aug 4. 63(3). 420-32. doi: 10.1016/j.molcel.2016.06.014.
- Londin E., Loher P., Telonis A.G. et al. Analysis of 13 cell types reveals evidence for the expression of numerous novel primate- and tissue-specific microRNAs. Proc. Natl. Acad. Sci USA. 2015 Mar 10. 112(10). E1106-15. doi: 10.1073/pnas.1420955112.
- Macias S., Cordiner R.A., Cáceres J.F. Cellular functions of the microprocessor. Biochem. Soc. Trans. 2013 Aug. 41(4). 838-43. doi: 10.1042/BST20130011.
- Medley J.C., Panzade G., Zinovyeva A.Y. microRNA strand selection: Unwinding the rule. WIREs RNA. 2020. https://doi.org/10.1002/wrna.1627.
- Mikkelsen T.S., Ku М., Jaffe D.B. et al. Genome-wide maps of chromatin state in pluripotent and lineage-committed cells. Nature. 2007 Aug 2. 448(7153). 553-60. Doi: 10.1038/nature06008.
- Nguyen T.A., Jo M.H., Choi Y.G. et al. Functional Anatomy of the Human Microprocessor. Cell. 2015 Jun 4. 161(6). 1374-87. doi: 10.1016/j.cell.2015.05.010.
- Ni W.J., Leng X.M. Dynamic miRNA-mRNA paradigms: New faces of miRNAs. Biochem. Biophys. Rep. 2015 Oct 28. 4. 337-341. doi: 10.1016/j.bbrep.2015.10.011.
- Nicholson A.W. Ribonuclease III mechanisms of double-stranded RNA cleavage. Wiley Interdiscip. Rev. RNA. 2014 Jan-Feb. 5(1). 31-48. doi: 10.1002/wrna.1195.
- Nuovo G., Amann V., Williams J. et al. Increased expression of importin-β, exportin-5 and nuclear transportable proteins in Alzheimer’s disease aids anatomic pathologists in its diagnosis. Ann. Diagn. Pathol. 2018 Feb. 32. 10-16. doi: 10.1016/j.anndiagpath.2017.08.003.
- Obsteter J., Dovc P., Kunej T. Genetic variability of microRNA regulome in human. Mol. Genet. Genomic. Med. 2015 Jan. 3(1). 30-9. doi: 10.1002/mgg3.110.
- Park J.E., Heo I., Tian Y. et al. Dicer recognizes the 5’ end of RNA for efficient and accurate processing. Nature. 2011 Jul 13. 475(7355). 201-5. doi: 10.1038/nature10198.
- Ratnadiwakara M., Mohenska M., Änkö M.L. Splicing factors as regulators of miRNA biogenesis — links to human disease. Semin. Cell. Dev. Biol. 2017 Oct 19. pii: S1084-9521(17)30124-6. doi: 10.1016/j.semcdb.2017.10.008.
- Robertson J.C., Jorcyk C.L., Oxford J.T. DICER1 Syndrome: DICER1 Mutations in Rare Cancers. Cancers (Basel). 2018 May 15. 10(5). pii: E143. doi: 10.3390/cancers10050143.
- Romero-Cordoba S.L., Salido-Guadarrama I., Rodriguez-Dorantes M., Hidalgo-Miranda A. miRNA biogenesis: biological impact in the development of cancer. Cancer Biol. Ther. 2014. 15(11). 1444-55. doi: 10.4161/15384047.2014.955442.
- Rostami Mogaddam M., Safavi Ardabili N., Shafaeei Y. et al. Overexpression of Drosha, DiGeorge syndrome critical region gene 8 (DGCR8), and Dicer mRNAs in the pathogenesis of psoriasis. J. Cosmet. Dermatol. 2017 Dec. 16(4). e48-e53. doi: 10.1111/jocd.12336.
- Song M.S., Rossi J.J. Molecular mechanisms of Dicer: endonuclease and enzymatic activity. Biochem. J. 2017 May 4. 474(10). 1603-1618. doi: 10.1042/BCJ20160759.
- Svobodova E., Kubikova J., Svoboda P. Production of small RNAs by mammalian Dicer. Pflugers. Arch. 2016 Jun. 468(6). 1089-102. doi: 10.1007/s00424-016-1817-6.
- Tan G.C., Dibb N. IsomiRs have functional importance. Malays J. Pathol. 2015 Aug. 37(2). 73-81.
- Tang R., Li L., Zhu D. et al. Mouse miRNA-709 directly regulates miRNA-15a/16-1 biogenesis at the posttranscriptional level in the nucleus: evidence for a microRNA hierarchy system. Cell. Res. 2012 Mar. 22(3). 504-15. doi: 10.1038/cr.2011.137.
- Tsutsumi A., Kawamata T., Izumi N. et al. Recognition of the pre-miRNA structure by Drosophila Dicer-1. Nat. Struct. Mol. Biol. 2011 Sep 18. 18(10). 1153-8. doi: 10.1038/nsmb.2125.
- Vogel T.W., Manjila S., Cohen A.R. Novel neurodevelopmental disorder in the case of a giant occipitoparietal meningoencephalocele. J. Neurosurg. Pediatr. 2012 Jul. 10(1). 25-9. doi: 10.3171/2012.3.PEDS11559.
- Wen J., Lv Z., Ding H. et al. Association of microRNA biosynthesis genes DROSHA and DGCR8 polymorphisms with cancer susceptibility: a systematic review and meta-analysis. Biosci. Rep. 2018 Apr 13. pii: BSR20180072. doi: 10.1042/BSR20180072.
- Wen J., Gao Q., Wang N. et al. Association of microRNA-related gene XPO5 rs11077 polymorphism with susceptibility to thyroid cancer. Medicine (Baltimore). 2017 Apr. 96(14). e6351. doi: 10.1097/MD.0000000000006351.
- Wilson R.C., Tambe A., Kidwell M.A. et al. Dicer-TRBP complex formation ensures accurate mammalian microRNA biogenesis. Mol. Cell. 2015 Feb 5. 57(3). 397-407. doi: 10.1016/j.molcel.2014.11.030.

/112.jpg)
/113.jpg)
/114.jpg)
/115.jpg)
/115_2.jpg)
/116.jpg)
/116_2.jpg)